Итак, дорогие читатели, в прошлом материале мы с вами выполнили оцифровку кривых Каплан-Мейера (общая выживаемость) из опубликованной научной статьи с результатами клинического исследования нусинерсена для терапии спинальной мышечной атрофией (https://www.nejm.org/doi/full/10.1056/nejmoa1702752). Для этого мы использовали специальную бесплатную утилиту WebPlotDigitizer (https://automeris.io/WebPlotDigitizer/). Теперь у нас есть Эксель файл с координатами кривых нусинерсена и плацебо (sham control), в который мы добавили информацию о временных отметках для которых есть данные о количестве пациентов под риском и, собственно, количество пациентов под риском в течение исследования. Вот этот файл Эксель:
Задачами этого второго материала является восстановление (имитация) индивидуальных данных пациентов в формате «время, статус, группа исследования» и выполнение регрессии по Коксу – вычисление Hazard Ratio (HR) на основании восстановленных данных и сравнение с опубликованным HR. Кроме того, восстановленные данные нам потребуются для третей части, в которой мы заглянем за горизонт клинического исследования и попробуем смоделировать в долгосрочном горизонте общую выживаемость.
Единственное «но», для того, чтобы выполнить все поставленные задачи нам будет недостаточно функционала, который предлагает Эксель. Нам потребуется программная среда R и программная оболочка R-Studio, которые являются бесплатными, но исключительно мощными инструментами, позволяющими решать практически все биостатистические задачи (meta-analysis, net meta-analysis, population-adjusted indirect comparisons, CEA, CUA и многое другое). Согласен, потребуется некоторое время для изучения R, но после этого ваши возможности в ОТЗ перестанут быть ограниченными функционалом Эксель, это будет качественное изменение, которые вы почувствуете сами и ваши коллеги.
Для быстрого погружения в R советую набрать в Гугл что-то вроде «R and R-Studio quick start», бесплатных материалов более, чем достаточно. В продолжении этого материала будет подразумеваться, что вы установили R и R-Studio, а также умеете выполнять простые действия в R – копировать код и запускать его. В тексте кода я буду давать #комментарии – описывать, что делает эта строка кода.
Что же, раз теперь нас ничего не держит, то вперед и с песнями. Для восстановления (имитации) пациентских данных мы будем использовать метод Patricia Guyot et al. (https://bmcmedresmethodol.biomedcentral.com/articles/10.1186/1471-2288-12-9). Работать будем с подключаемой библиотекой IPDfromKM — reconstruct individual patient data from published Kaplan-Meier survival curves, созданной Na Liu et al. (https://bmcmedresmethodol.biomedcentral.com/articles/10.1186/s12874-021-01308-8 ).
Устанавливаем и подключаем необходимые библиотеки:
install.packages("IPDfromKM") #устанавливаем библиотеку для восстановления индивидуальных данных о пациентах
install.packages("survminer") #устанавливаем библиотеку для статистического анализа выживаемости
install.packages("readxl") #устанавливаем библиотеку для загрузки Эксель файлов в R
library(IPDfromKM) #подключаем библиотеку
library(survminer) #подключаем библиотеку
library(readxl) #подключаем библиотеку
Загружаем подготовленные данные из файла Эксель «dataset.xlsx»:
dataset <- read_excel("D:/Extractor/dataset.xlsx") #загружаем данные в переменную dataset
View(dataset) #проверяем, что загрузили
Прежде, чем следовать далее, договоримся о следующем, arm0 – означает контрольную группу (в данном случае группа плацебо), arm1 – эффективную группу (в данном случае группа нусинерсена).
trisk <- dataset$trisk #переменная trisk будет содержать информацию о временных отметках, где нам известно количество пациентов под риском
trisk <- trisk[1:6]
trisk
[1] 0 13 26 39 52 56
nrisk_0 <- dataset$nrisk_arm0 #переменная nrisk_0 будет содержать данные о количестве пациентов под риском в контрольной группе
nrisk_0 <- nrisk_0[1:6]
nrisk_0
[1] 41 33 23 17 12 10
nrisk_1 <- dataset$nrisk_arm1 #переменная nrisk_1 будет содержать данные о количестве пациентов под риском в группе нусинерсена
nrisk_1 <- nrisk_1[1:6]
nrisk_1
[1] 80 71 58 41 28 23
Далее с помощью функции preprocess библиотеки IPDfromKM выполняем предварительную обработку полученных при оцифровке координат. Алгоритм очищает данные от парадоксальных координат, каждая последующая точка Х не может иметь значение большее значение Y. Более подробное описание этого этапа вы найдете по ссылке в начале этой статьи.
pre_arm_0 <- preprocess(dat = dataset[,3:4], #загружаем данные для контрольной группы (3 и 4 столбец в таблице)
trisk = trisk, #время, где известно количество пациентов под риском. Если в публикации нет таких данных – можно не указывать.
nrisk = nrisk_0, #количество пациентов под риском. Если в публикации нет таких данных – можно не указывать.
maxy = 1) #максимальное значение по оси Х (у нас 1, но может быть 100%, тогда указывается = 100)
Выполняем аналогичное упражнение для группы нусинерсена.
pre_arm_1 <- preprocess(dat = dataset[,1:2],
trisk = trisk,
nrisk = nrisk_1,
maxy = 1)
И, наконец то, восстанавливаем индивидуальные данные о пациентах.
est_arm_0 <- getIPD(prep = pre_arm_0, #объект, полученные в результате препроцессинга данных группы плацебо
armID = 0, #как договорились раньше, 0 – контрольная группа, 1 - эффективная
tot.events = 41-25) # количество событий, в данном случае смертей, берем из публикации: 41 – количество пациентов в группе минус 25 – количество выживших пациентов. Если в публикации нет таких данных – можно не указывать
Выполняем аналогичное упражнение для группы нусинерсена.
plot(est_arm_0)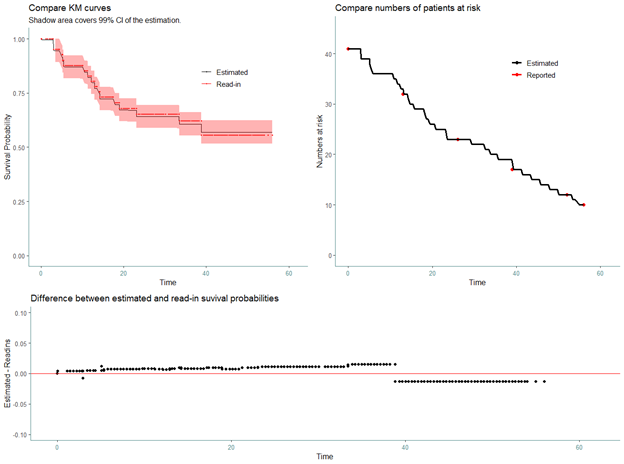
И для группы нусинерсена.
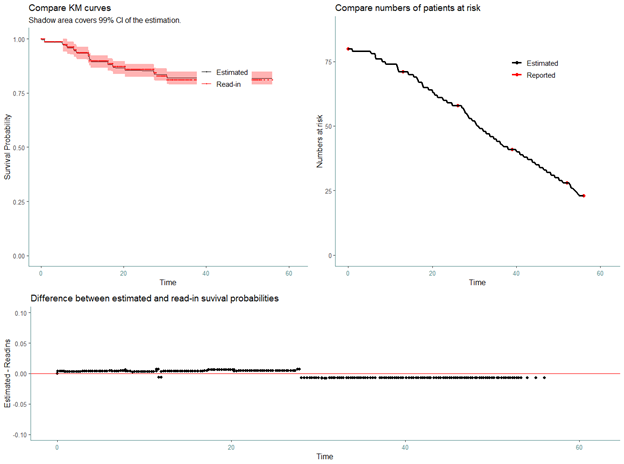
Наблюдаем некоторые расхождения между восстановленными данными и координатами, которые мы получили при оцифровке. Не будем погружаться в детали, будем считать их незначительными в рамках наших задач.
Переходим к финальному этапу наших упражнений, готовим набор данных для регрессии по Коксу и вычисления HR (hazard ratio).
data_for_cox <- rbind(est_arm_0$IPD, est_arm_1$IPD) #объединяем восстановленные в одну таблицу
cox_regr <- coxph(Surv(data_for_cox$time, data_for_cox$status) ~ data_for_cox$treat) #выполняем регрессию по Коксу с одним модификаторм – терапия или отсутствие терапии
summary(cox_regr) # оцениваем результаты

Hazard ratio – exp(coef) = 0,3765
95% доверительный интервал: 0,1855 (lower .95) – 0,764 (upper .95)
Значение р – Pr(>|z|) = 0.00682
Ниже приведена сравнительная таблица (est – estimated, на основании восстановленных данных).
HR for death
| HR | HR est | LB | LB est | UB | UB est | p | p est |
| 0,37 | 0,3765 | 0,18 | 0,1855 | 0,77 | 0,764 | 0,004 | 0,00682 |
Как мы видим, полученные значения очень близки к опубликованным. На данном этапе я удовлетворен полученными результатами, которые были получены всего лишь из одной картинки.
Напишите в комментариях, какие результаты получились у вас, если вы прошли все этапы самостоятельно. Напоследок, создадим красивый график кривых Каплана-Мейера на основании восстановленных данных.
ggsurvplot(
surv_fit_for_plot, # survfit object with calculated statistics.
data = data_for_cox,# data used to fit survival curves.
risk.table = TRUE, # show risk table.
xlim = c(0, 56),
pval = FALSE, # show p-value of log-rank test.
conf.int = T, # show confidence intervals for
palette = "aaas",
xlab = "Weeks", # customize X axis label.
ylab = "Overall survival",
censor = F,
break.time.by = 13, # break X axis in time intervals by 13.
ggtheme = theme_light(), # customize plot and risk table with a theme.
risk.table.y.text.col = T,# color risk table text annotations.
risk.table.height = 0.25, # the height of the risk table
risk.table.y.text = T,# show bars instead of names in text annotations
# in legend of risk table.
ncensor.plot = FALSE, # plot the number of censored subjects at time t
ncensor.plot.height = 0.25,
conf.int.style = "step", # customize style of confidence intervals
surv.median.line = "hv", # add the median survival pointer.
legend.title = "",
legend.labs =
c("control", "nusinersen")# change legend labels.
)
График, созданный на восстановленных данных
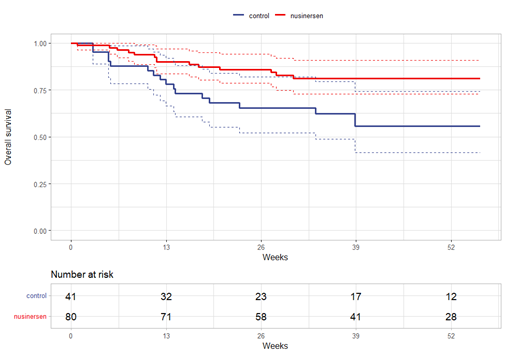
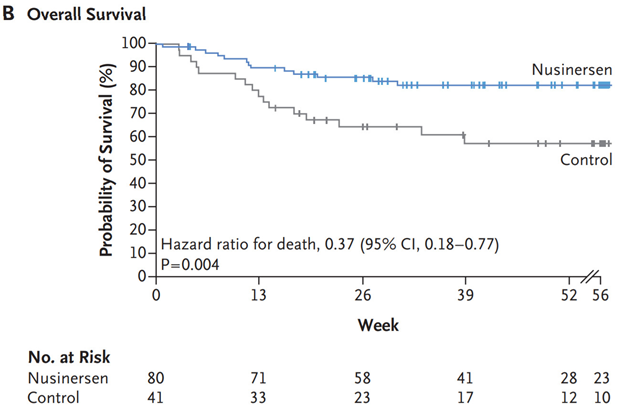
График из статьи с результатами исследования
В следующем материале, но основании полученных данных мы заглянем чуть дальше за горизонт наблюдения клинического исследования и попробуем создать параметрическую модель долгосрочной общей выживаемости. Там же, вы найдете файл с R кодом этого материала.
Дорогой читатель, буду рад твоим комментариям, предложениям и вопросам!